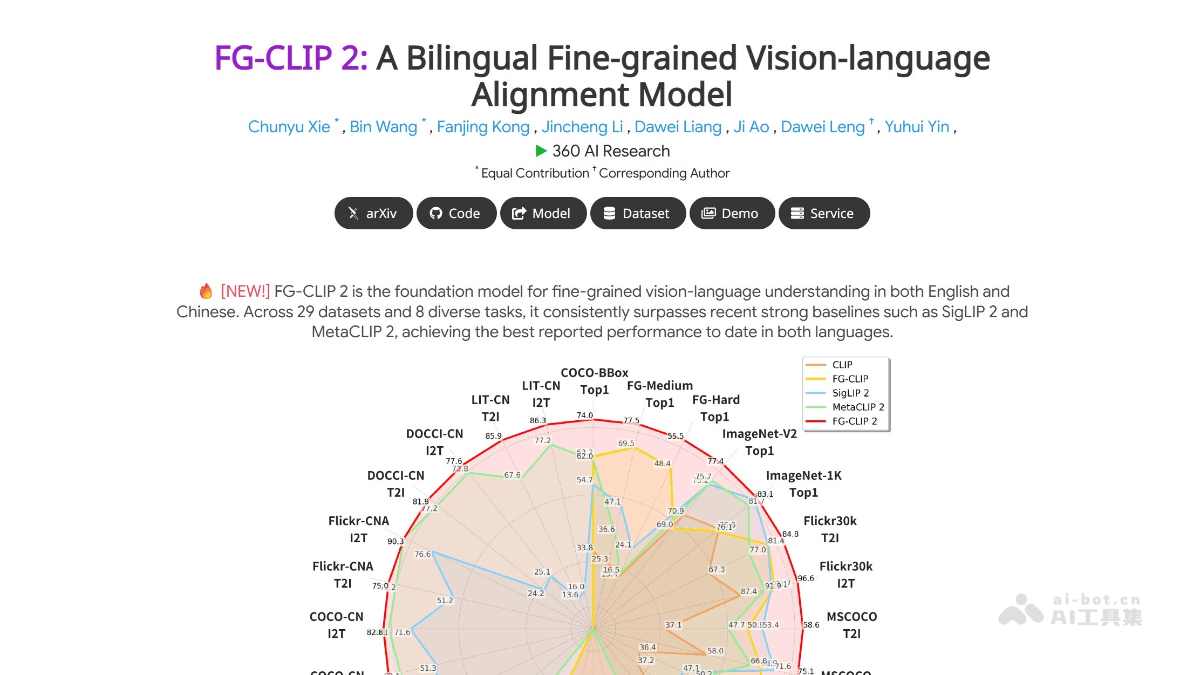
人工智能(AI)和机器学习在科学领域,特别是在生物学中,取得了诸多引人瞩目的成就。例如,它们在设计能分解塑料的酶,或是开发能阻断蛇毒的蛋白质方面,都展现了卓越的能力。在当前AI炒作甚嚣尘上的时代,人们很容易产生一种错觉,认为只要将AI应用于海量生物数据,便能一劳永逸地解决大多数生物学问题,从而规避大量繁琐的实验乃至动物研究的伦理困境。然而,生物学的复杂性远超简单的蛋白质结构分析。因此,当前断言AI能够同等有效地处理生物学的所有方面,显然为时过早。近期一系列关于基因活性的研究报告,揭示了当前AI工具在某些特定生物学任务上的局限性,这无疑为过度的AI乐观主义敲响了警钟。
AI与基因活性的复杂性
来自海德堡的三位研究人员——康斯坦丁·阿曼-埃尔策(Constantin Ahlmann-Eltze)、沃尔夫冈·胡贝尔(Wolfgang Huber)和西蒙·安德斯(Simon Anders)开展了一项引人深思的研究,旨在评估一组旨在预测细胞在不同条件下基因活性变化的AI软件。他们的发现与其他同期发表的研究不谋而合,共同指向了一个核心结论:在预测基因活性方面,目前的AI系统表现平平,甚至未能超越一些经过刻意简化的预测方法。
基因活性是生命活动的基础。虽然每个细胞都携带了人类基因组中大约2万个基因的副本,但在特定细胞中,并非所有基因都处于活跃状态——“活跃”在此指它们正在产生信使RNA(mRNA)。有些基因编码的蛋白质承担着细胞的基本功能,因此始终保持高水平活性;另一些则仅在特定细胞类型中活跃,如神经细胞或皮肤细胞;还有些基因只在特定环境条件下被激活,如缺氧或高温。多年来,科学家们已进行了大量研究,在不同条件下检查特定细胞类型中所有基因的活性。这些研究涵盖了从使用基因芯片检测细胞群中存在的mRNA,到测序单个细胞中分离出的RNA并利用数据识别活跃基因等多种方法。这些累积的数据,尽管可能不完整,但共同描绘了一幅基因活性与不同生物学环境之间联系的广阔图景。理论上,这幅图景可以用于训练AI,使其能够预测在尚未测试的条件下基因的活性。
AI模型表现不佳的深层原因
阿曼-埃尔策、胡贝尔和安德斯测试了一组“单细胞基础模型”(single-cell foundation models),这些模型已经通过上述基因活性数据进行了训练。“单细胞”部分表明这些模型基于个体细胞而非细胞类型平均值获取的基因活性数据进行训练。而“基础模型”则意味着它们在大量多样化数据上进行了预训练,但需要针对特定任务进行额外的微调。
本研究中的核心任务是预测基因改变后基因活性可能如何变化。当单个基因缺失或激活时,有时只会影响该基因产生的信使RNA。然而,有些基因编码的蛋白质可以调节一系列其他基因,这种情况下,可能会导致数十个基因的活性发生变化。在其他情况下,基因的缺失或激活可能会影响细胞代谢,进而导致基因活性的广泛改变。
更复杂的情形出现在两个基因参与的相互作用中。在许多情况下,这两个基因的功能可能不相关,因此其效应表现为简单的叠加:一个基因缺失引起的变化,加上另一个基因缺失引起的变化。但如果它们的功能存在某种程度的重叠,就可能出现某些变化的增强、另一些变化的抑制,以及其他意想不到的复杂变化,即所谓的“协同效应”。
为了探索这些效应,研究人员利用CRISPR DNA编辑技术,有目的地改变了一个或多个基因的活性,然后对细胞中的所有RNA进行测序,以观察发生了何种变化。这种方法(称为Perturb-seq)非常有用,因为它能让我们了解被改变的基因在细胞中的作用。对于阿曼-埃尔策、胡贝尔和安德斯而言,它提供了他们所需的数据,以确定这些基础模型是否能够被训练来预测其他基因活性的后续变化。
研究人员首先对基础模型进行了进一步训练,使用了通过CRISPR激活一个或两个基因的实验数据。训练数据集包含了100个单独基因激活的数据和62个双基因激活的数据。随后,研究人员要求AI软件包预测另外62对被激活基因的实验结果。作为对比,研究人员还使用了两个极其简单的模型进行预测:一个模型始终预测不会发生任何变化,另一个模型则始终预测累加效应(即激活基因A和基因B将产生激活A引起的变化加上激活B引起的变化的总和)。
结果令人失望。研究人员总结道:“所有模型的预测误差都显著高于累加基线模型。”即使研究人员使用其他衡量AI预测准确性的指标,这一结果也依然成立。问题似乎在于,经过训练的基础模型在预测双基因改变何时会产生复杂变化模式(即一个基因的改变与第二个基因的改变发生协同作用)方面表现不佳。“深度学习模型很少能预测出协同相互作用,而这些预测的正确率则更低。”研究人员总结道。在专门针对这些基因间协同作用的独立测试中,结果显示,没有任何一个模型的表现优于始终预测无变化的简化系统。
生物学复杂性下的AI发展路径
这项研究的总体结论非常明确。研究人员指出:“由于我们刻意简化的基线模型无法代表现实的生物复杂性,但却并未被基础模型超越,我们因此得出结论,后者旨在提供细胞状态的可泛化表示并预测尚未进行实验结果的目标,仍然遥不可及。”
需要强调的是,“仍然遥不可及”并不意味着我们永远无法开发出能解决这一问题的AI。同样,这也不意味着这些结论适用于所有细胞状态(研究结果特指基因活性),更遑论适用于整个生物学领域。然而,在当前人们对AI在少数领域取得成功而普遍认为其即将无所不能的过度热情中,这项研究无疑提供了一个宝贵的警示。
生物系统的内在复杂性,特别是基因调控网络的高度非线性、动态变化以及多种环境因素的交互影响,使得简单的线性叠加或基于有限数据模式的识别难以捕捉其全貌。传统的机器学习模型往往在处理这种高维、非线性和高度相互关联的数据时面临挑战,尤其是在数据稀疏或相互作用模式新颖的情况下。未来的AI研究可能需要更深入地结合生物学的先验知识,开发能够理解和模拟生物学原理的“知识增强型”AI模型,而不仅仅是依赖于纯粹的数据驱动模式识别。例如,将基因调控网络的拓扑结构、信号转导通路的时序逻辑等生物学理论融入到模型架构中,或许能帮助AI更好地捕捉复杂相互作用。
此外,当前单细胞测序技术的飞速发展虽然提供了前所未有的数据粒度,但也带来了新的挑战,例如数据噪声、批次效应以及如何有效整合不同类型、不同来源的单细胞数据。构建真正能够泛化并准确预测未知生物学状态的基础模型,需要克服这些技术和计算上的瓶颈。这可能涉及开发新的数据归一化、整合方法,以及设计更具鲁棒性和可解释性的模型架构。
具体而言,要让AI在基因活性预测上取得突破,可能需要从以下几个方面着手:
数据质量与规模的提升:尽管已有大量基因活性数据,但高维度、低噪声、跨物种、跨条件、涵盖更多复杂扰动组合的标准化数据集仍然是AI学习的关键。同时,开发更高效的单细胞多组学数据整合技术,将基因组、转录组、蛋白质组甚至代谢组信息结合起来,为AI提供更全面的细胞状态视图。
更复杂的模型架构:目前的“基础模型”可能在捕捉简单的统计关联方面表现良好,但在理解生物学中普遍存在的非线性、反馈回路和多层次调控机制上仍显不足。未来需要探索结合图神经网络(Graph Neural Networks)来建模基因间的相互作用网络,或者利用因果推理模型来识别真正的驱动因素而非仅仅是相关性。同时,发展能够融入生物学先验知识(如通路信息、蛋白质相互作用数据)的物理启发式AI模型,将有望提升其预测的准确性和可解释性。
可解释性与鲁棒性:当前的深度学习模型往往被诟病为“黑箱”,难以解释其预测的依据。在生物学这样高风险的领域,理解AI为何做出特定预测至关重要。因此,发展可解释AI(XAI)技术,让模型不仅能预测,还能指出哪些基因、哪些通路对预测结果贡献最大,将有助于科学家验证模型、发现新的生物学机制。同时,提升模型对噪声、异常数据和未见过的实验条件的鲁棒性,也是其能在实际研究中广泛应用的前提。
跨学科深度融合:这不仅仅是让生物学家提供数据给AI专家,而是需要生物学家、计算科学家、统计学家甚至物理学家共同协作,从生物学问题本身出发,设计出更符合生物学逻辑、更能捕捉生物系统本质特征的AI模型。例如,在理解基因协同效应时,可能需要生物学家提出具体的假设,AI模型则负责验证这些假设并在大规模数据中发现新的模式。
综上所述,虽然AI在生物学的特定领域展现了巨大潜力,但在理解并预测基因活性等深层、复杂的生物学机制方面,我们仍处于探索阶段。未来的发展需要更优质的数据、更智能的模型、更强大的计算能力以及更紧密的跨学科合作。只有这样,AI才能真正成为揭示生命奥秘的强大工具,而非仅限于处理表面数据的速效药。